

我是杜克大学分子遗传学与微生物学系Lawrence David教授实验室里的一名博士研究生。我对微生物之间的"社交生活"非常感兴趣,包括细胞层面的微观互动,以及复杂菌群生态系统中的群体交互。在我过去的课题里,我研究过细菌是如何利用分子武器"互殴"消灭对方的,也研究过癌症病人的饮食习惯对他们的肠道菌群以及治疗效果有着怎样的影响。在过去9年的科研经历里,我的时间大致六四开:60%在实验台前做实验,40%则对着电脑跑代码、分析数据、画图。
最近我正在尝试把AI智能体、MCP协议、自动化工作流,以及vibe coding这类新工具融入我的日常工作,以优化我的项目管理和数据分析流程。对我来说,AI工具是提升科研学习效率和工作效能的加速器,但我个人并不支持或使用AI生成式艺术。我论文中的图表和示意图均由我本人使用R或者BioRender手工绘制。
欢迎通过电子邮件或LinkedIn与我联系。我完整的发表记录可在Google Scholar上查阅。我的论文中用于分析与数据可视化的代码则发布在我的GitHub上。
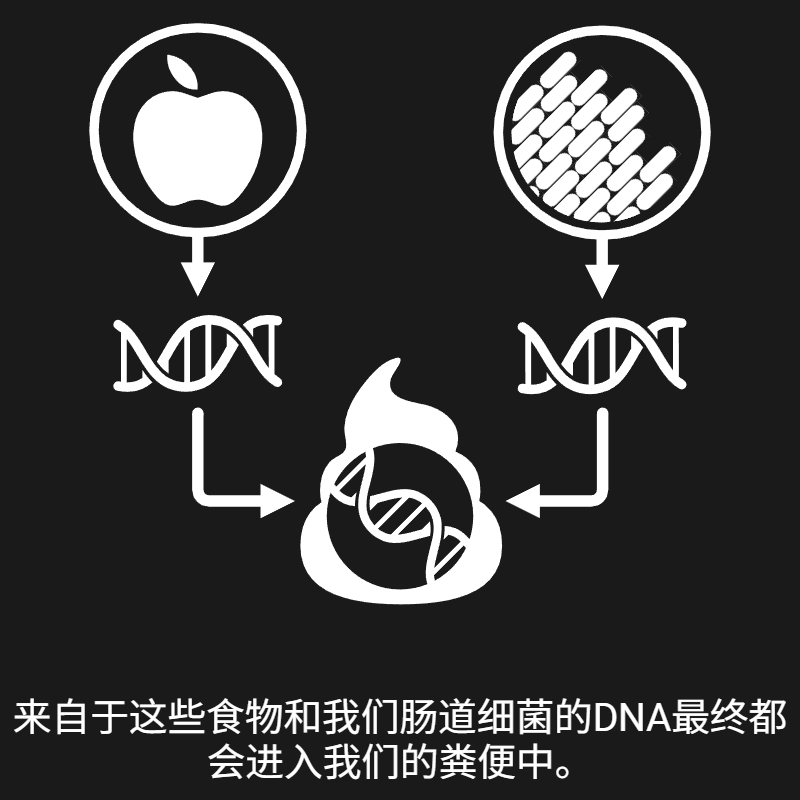
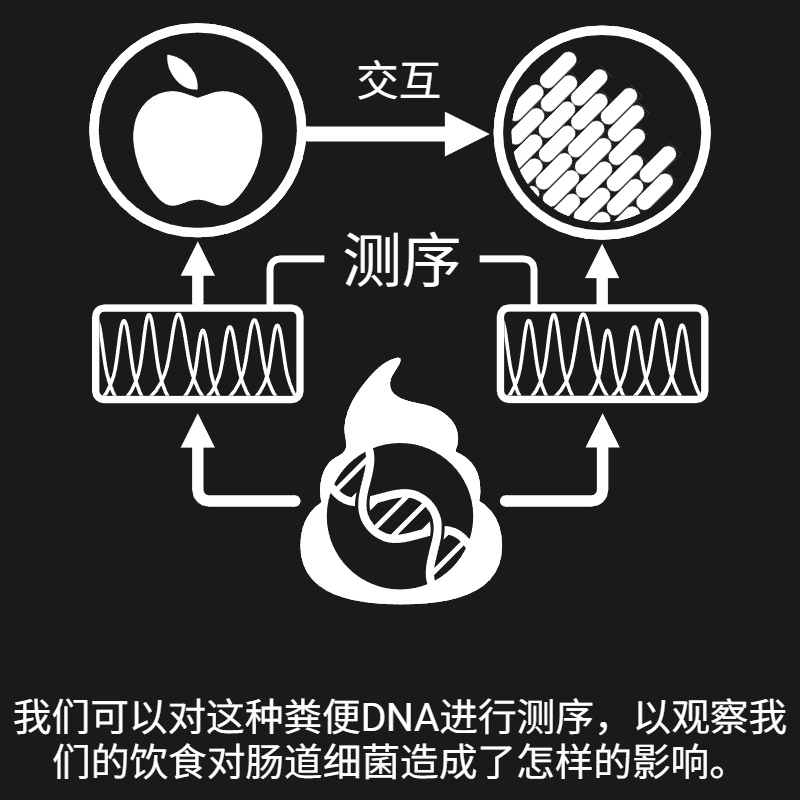
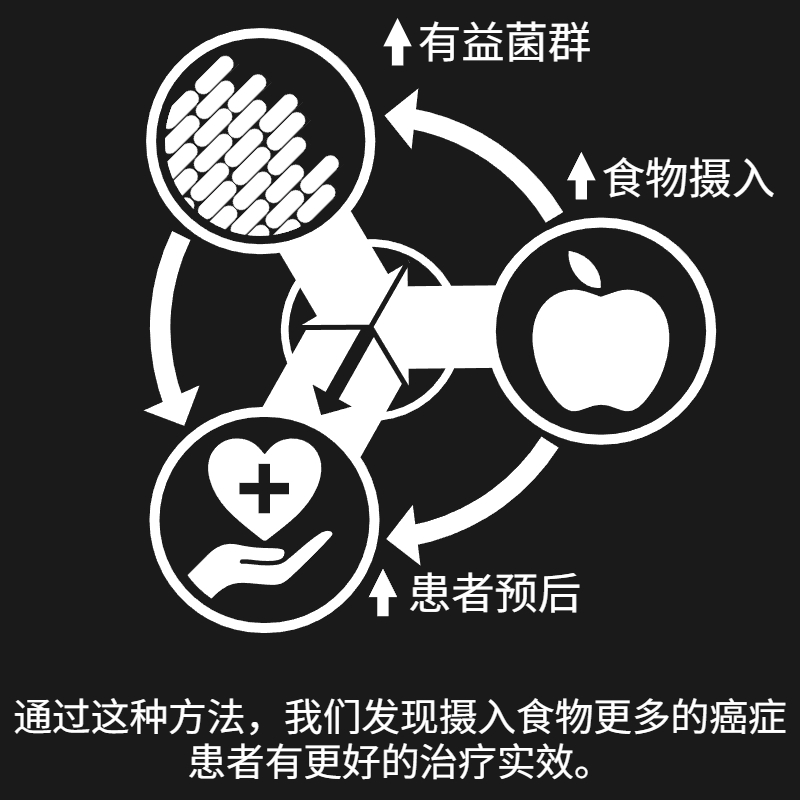
我的研究围绕两个核心问题展开:1)微生物是如何与其环境中的不同要素相互作用的;2)我们如何利用这些相互作用来改善人类健康。我一直在追寻这两个问题的答案, 而以下是我目前的一些发现:





我的科研经历让我在实验 (湿实验) 和计算 (干实验) 两方面都积累了丰富的技能,具体清单列在右侧。其中,浅灰色标注的是我用过但尚未出现在正式发表成果中的工具或技能。
Zeng J., García-González A. P., Epstein P., Bauer A. E., Jiang S., Kirtley M. C., Neubert B. C., Rivera C. N., Bergens M. A., Bush A. T., Hill L., Gauthier J., McGriff C., Tang H., Andermann T. M., Jobin C., Chao N. J., Dahl W. J., Wingard J. R., Sung A. D., & David L. A. (2025). Dietary signatures from fecal DNA predict hematopoietic stem cell transplantation outcomes. 已提交。
Aqeel A., Kay M. C., Zeng J., Petrone B. L., Yang C., Truong T., Brown C. B., Jiang S., Carrion V. M., Bryant S., Kirtley M. C., Neshteruk C. D., Armstrong S. C., & David L. A. (2025). Grocery intervention and DNA-based assessment to improve diet quality in pediatric obesity: a pilot randomized controlled study. Obesity (Silver Spring, Md.), 33(2), 331–345. [链接]
Letourneau J., Carrion V. M., Zeng J., Jiang S., Osborne O. W., Holmes Z. C., Fox A., Epstein P., Tan C. Y., Kirtley M., Surana N. K., & David L. A. (2024). Interplay between particle size and microbial ecology in the gut microbiome. The ISME journal, 18(1), wrae168. [链接]
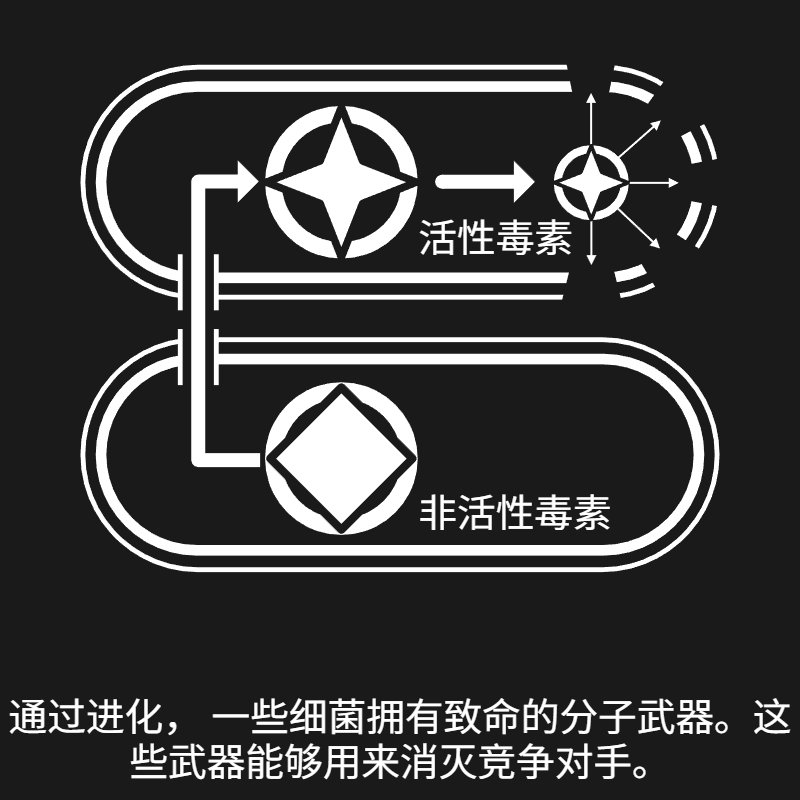
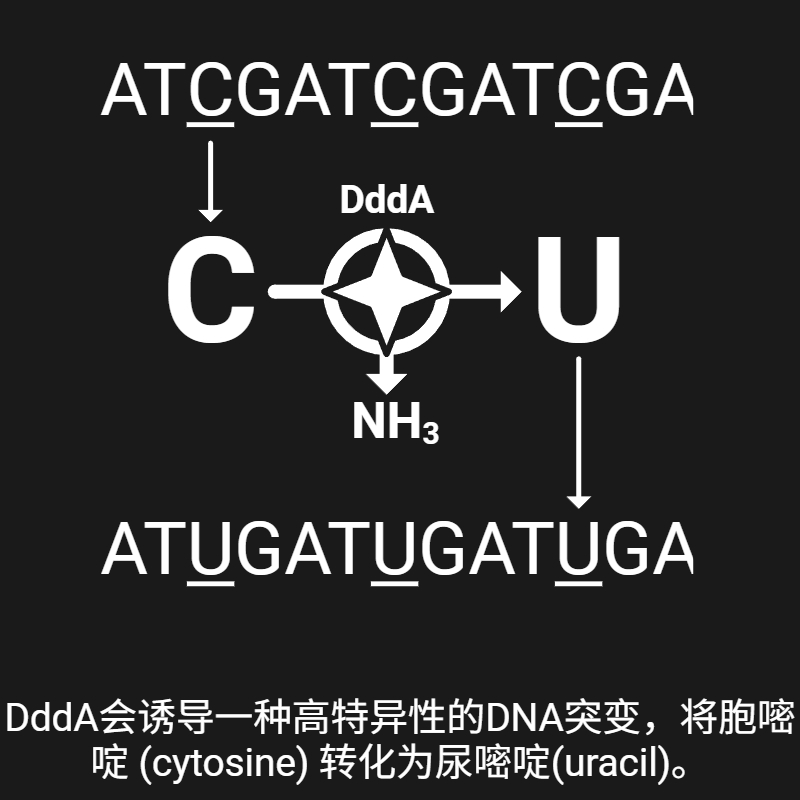
de Moraes M. H., Hsu F., Huang D., Bosch D. E., Zeng J., Radey M. C., Simon N., Ledvina H. E., Frick J. P., Wiggins P. A., Peterson S. B., & Mougous J. D. (2021). An interbacterial DNA deaminase toxin directly mutagenizes surviving target populations. eLife, 10, e62967. [链接]
Mok B. Y., de Moraes M. H., Zeng J., Bosch D. E., Kotrys A. V., Raguram A., Hsu F., Radey M. C., Peterson S. B., Mootha V. K., Mougous J. D., & Liu D. R. (2020). A bacterial cytidine deaminase toxin enables CRISPR-free mitochondrial base editing. Nature, 583(7817), 631–637. [链接]
我为已发表作品制作的一些图表示例。
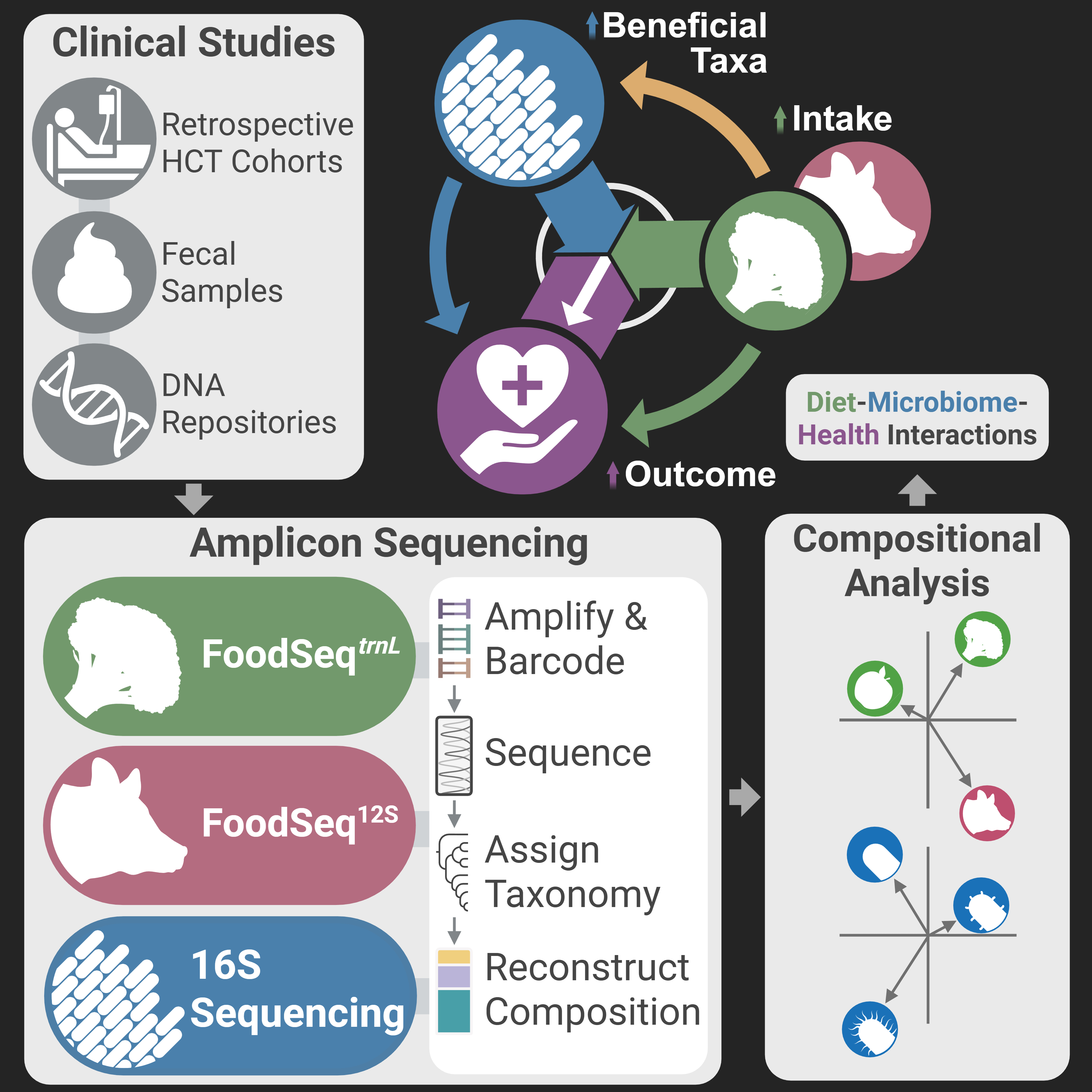
本图示总结了我的学位论文项目,整合了我们对植物trnL、动物12S和细菌16S标记基因的扩增子测序结果——所有数据均来自仅15毫克原始粪便样本提取的同一份DNA。
每个数据点代表一份粪便样本,不同的颜色表示该样本相对于骨髓移植的采集周数。双击图例中的标签可筛选样本。
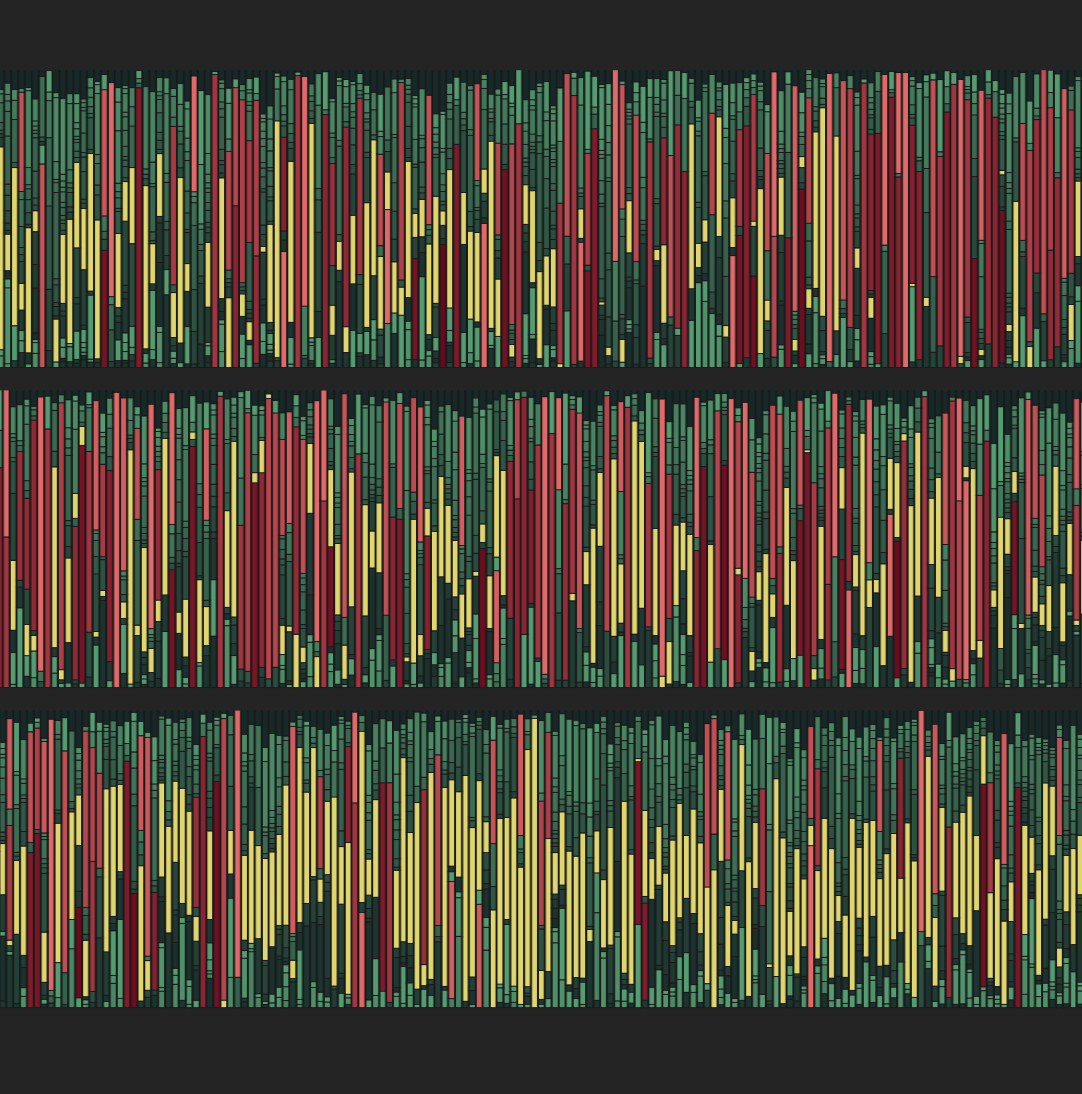
每个条形图代表一份粪便样本的微生物组组成。基于16S rRNA测序结果:黄色为Blautia,红色为相对丰度≥30%的非Blautia物种,绿色为相对丰度<30%的任何物种。
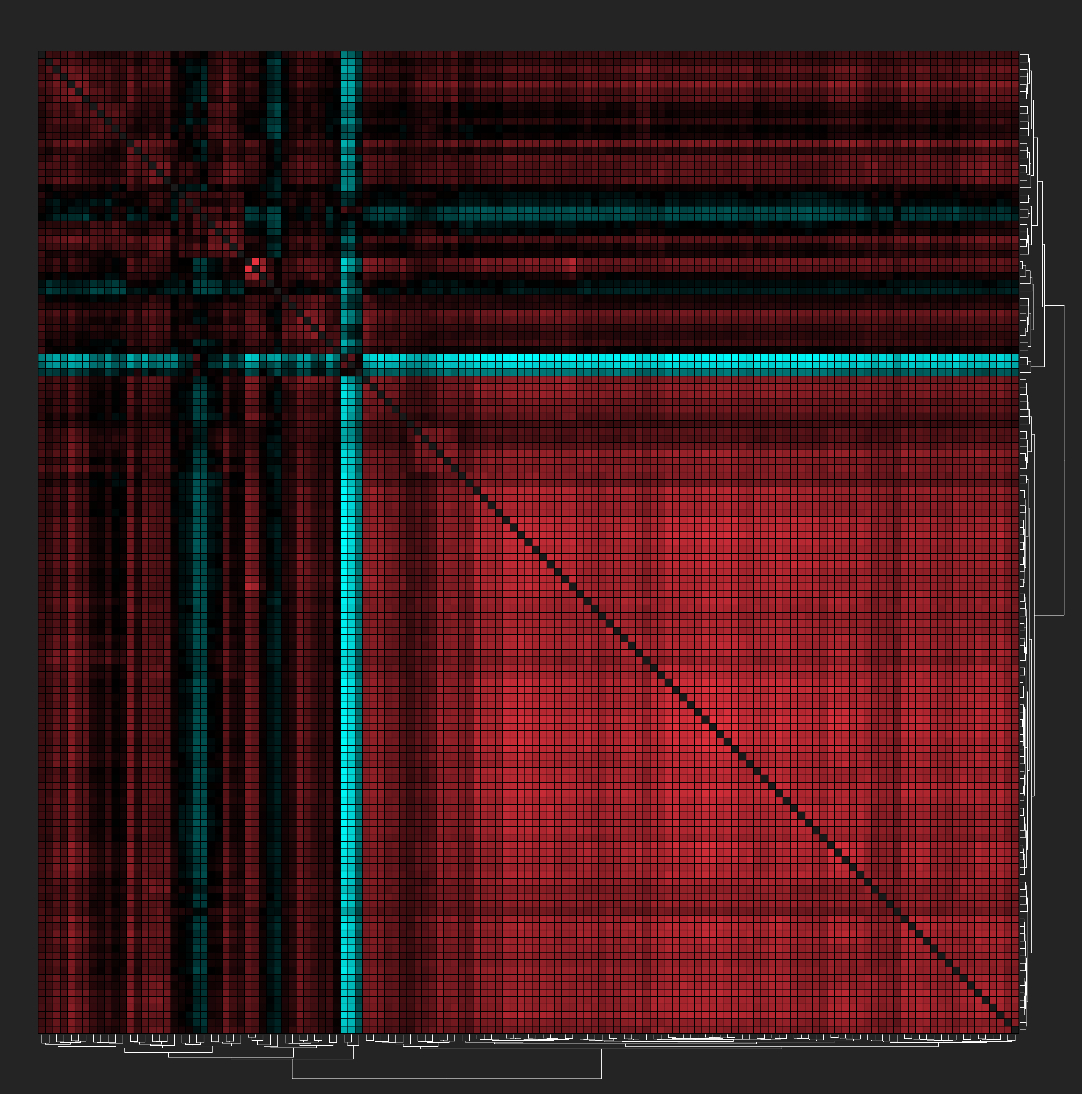
该热图展示了基于细菌丰度计算的欧氏距离矩阵:红色表示较低的距离,青色表示较高的距离。三种占主导地位的微生物分类群与几乎所有其他微生物类群均呈负相关。
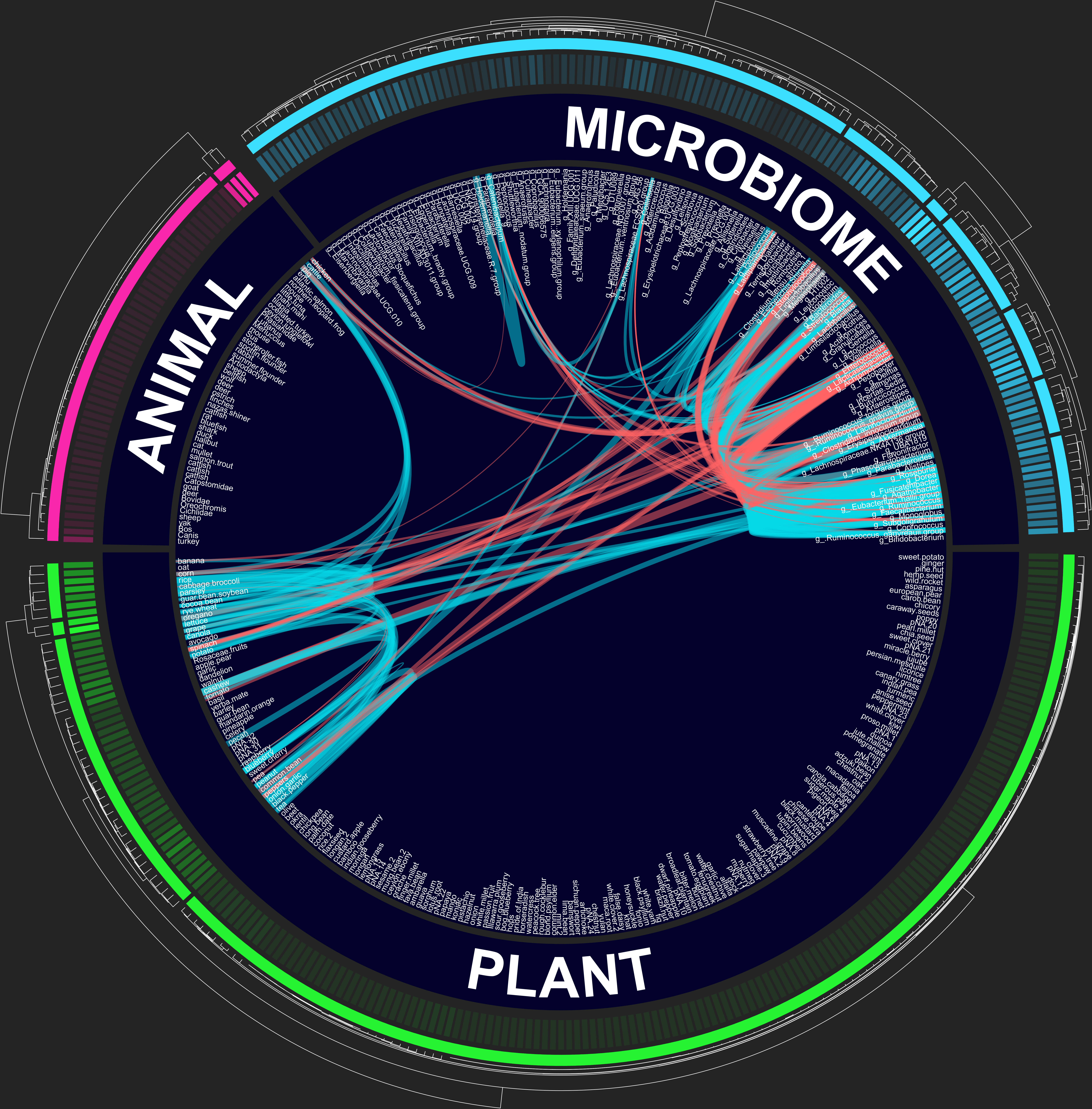
这些关联在食物类群之间或细菌类群内部更为明显,而不同数据集 (如食物与细菌) 之间的关联则较为罕见。这可能是因为饮食具有更强的瞬时性和动态性,而细菌组成在较长时间内可能更为稳定。